Novel deep learning model for moreaccurate prediction of drug-drug interaction effects
本篇推文引自:Novel deep learning model for more accurate prediction of drug-drug interaction effects
1. 摘要
背景:准确地预测药物-药物相互作用(DDIs)的影响对于更安全更有效的药物联合处方很重要。许多预测DDIs效应的计算方法已经被提出,目的是减少在体内或体外识别这些相互作用的难度,但预测性能仍有改进的空间。
结果:在本研究中,我们提出了一种新的深度学习模型来更准确地预测DDIs的效果。提出的模型使用自动编码器和深度前馈网络来预测DDIs的药理作用。深度前馈网络使用已知药物对的结构相似图谱(SSP)、基因本体(GO)术语相似图谱(GSP)和目标基因相似图谱(TSP)进行训练。结果表明,GSP和TSP都提高了单独使用SSP进行训练时的预测精度,并且自动编码器比主成分分析能更有效地进行降维。我们的模型比现有的方法表现出更好的性能,并确定了一些由医学数据库或现有研究支持的新型DDIs。
结论:我们提出了一种新的深度学习模型,可以更准确地预测DDIs及其作用,这可能有助于未来的研究发现新的DDIs及其药理作用。
2. 背景
联合药物治疗正成为治疗包括癌症、高血压、哮喘和艾滋病在内的多种疾病的一种有效的方法,因为它们可以提高药物疗效、降低药物毒性或降低个体耐药性。然而,药物联合使用可能导致药物间相互作用(DDIs),这是药物不良事件(ADEs)的主要原因。据估计,DDIs与所有报道的ADEs中30%的有关。此外,由于严重的DDIs导致的不良反应已经导致药物从市场上撤出。因此,准确预测DDIs的疗效对于患者更安全、更好的处方具有重要意义。
为了提高本研究的分类精度,我们提出了一种新的基于深度学习的模型,该模型使用了来自靶标基因及其已知功能的额外特征。我们构建了靶标相似性概要(TSP)、基因本体(GO)术语相似性概要(GSP)和SSP。因为在组合TSP、GSP和SSP时,输入大小太大,所以我们使用自动编码器来减少该特性。我们的自编码器模型训练最小化输入与输出的差值,同时训练最小化DDI标签的预测误差。
3. 结果
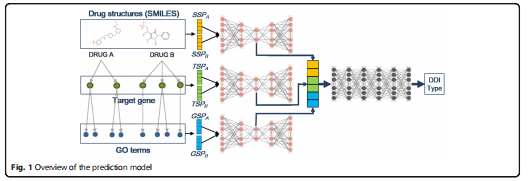
我们开发了一种新的深度学习模型来预测DDIs的药理作用。该模型使用一个自动编码器来减少药物对的三个相似度概要的维数,并使用一个深度前馈网络来预测DDI类型。利用已知药物对的化学结构(SSP)、靶基因(TSP)和靶基因的生物学/分子功能(GSP)计算出三种相似性。整个过程如图1所示,方法部分给出了详细的描述。
为了训练我们的模型,我们从DrugBank下载了已知177种DDIs的396,454个,以及药物的smile和靶基因信息。从BioGrid下载功能交互(FI)网络。FI网络由22,032个基因组成。GO数据库从Gene Ontology Consortium下载。GO数据库由45,106个GO词条组成,我们在生物过程中使用了29,692个GO词条。排除无目标基因信息的药物,排除DDIs小于5个的DDI类型。最后,实验使用了106种188,258种DDIs和1597种药物。
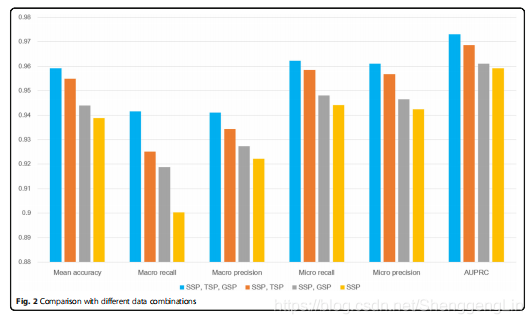
 我们的模型是使用SSP、TSP和GSP的不同组合来学习的。采用5倍交叉验证方法计算了准确度、宏观精度、宏观召回率、微观精度、微观召回率以及精度/召回曲线下的面积。这些性能指标如下:
我们的模型是使用SSP、TSP和GSP的不同组合来学习的。采用5倍交叉验证方法计算了准确度、宏观精度、宏观召回率、微观精度、微观召回率以及精度/召回曲线下的面积。这些性能指标如下: 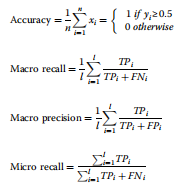 其中,n和l分别为样本数量和DDI类型,yi为样本i的DrugBank数据库中DDI真类型的预测值,TP、TN、FP和FN分别为真阳性、真阴性、假阳性和假阴性。
其中,n和l分别为样本数量和DDI类型,yi为样本i的DrugBank数据库中DDI真类型的预测值,TP、TN、FP和FN分别为真阳性、真阴性、假阳性和假阴性。 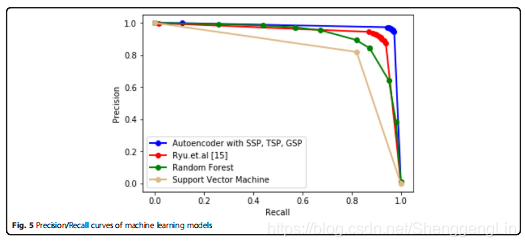
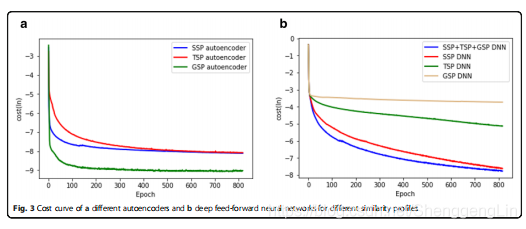
图2显示,加入TSP和GSP可以提高分类精度。仅使用GSP和TSP的测试和同时使用GSP和TSP的测试都没有产生良好的分类准确率(< 0.5)。我们还可以观察到,就AUPRC而言,TSP和GSP提高了分类精度。图3为自编码器和深度前馈网络的代价曲线,可以看出,TSP和GSP的深度前馈网络收敛时,代价较大。虽然GSP和TSP不是很好的单一相似度度量,但它们使用SSP提高了预测性能。
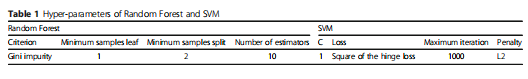
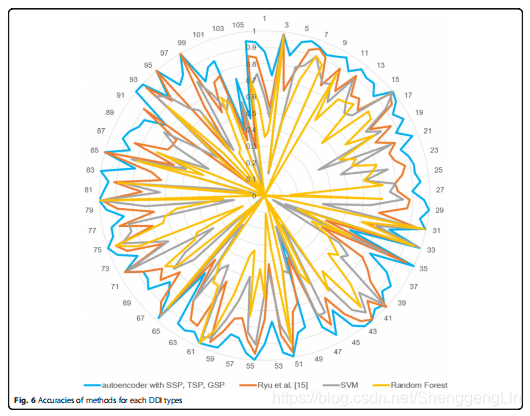
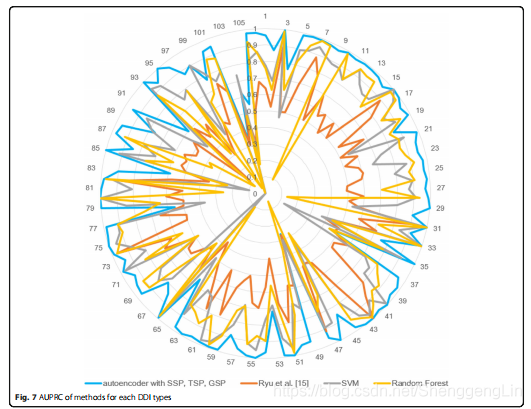
我们可以看到,使用自动编码器的SSP(图2中的黄色)产生的结果优于图4和图5中使用PCA的SSP。我们还可以证实,所提出的模型比SVM或随机森林等基线方法表现出更好的性能。SVM和Random Forest的超参数如表1所示。对于所提出的模型和图2、4、5中Ryu等的模型,使用自动编码器或主成分分析将特征数量减少到200个,而SVM和随机森林的特征没有减少。为了更具体地观察每种方法的性能,我们比较了每种DDI类型的结果。在使用本文模型的两种情况下,106种DDI类型中有101种的分类精度更高或相同(图6和图7)。
4. 讨论
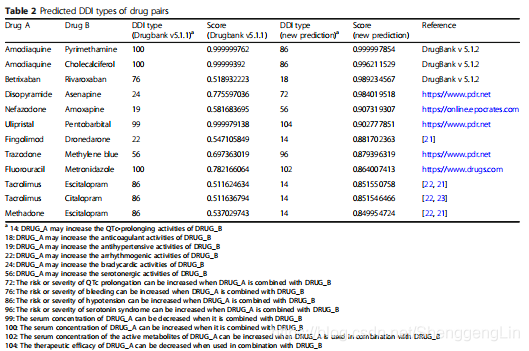
在5倍交叉验证结果的真实预测中,我们选择了DDI类型的预测值大于或等于0.5的药物对。在580对药物中,有86对(14.8%)药物得到了其他数据库或已有研究的支持。在86对支持的药物中,我们在表2中显示了12对预测评分为> 0.8的药物。Table 2中前3个DDIs的类型在DrugBank v5.1.1中为100,100和76,但在DrugBank v5.1.2中更新为86、86和18,我们对这3个DDIs的预测得分非常高。
我们的工作有两个潜在的局限性。首先,DrugBank中的DDIs多为推测的药代动力学相互作用,因此需要验证所提出模型预测的DDIs及其临床结果。其次,我们的设置通过迭代实验得到学习率、隐藏单位/层数、退出率等超参数的最优值,从而可以根据不同的数据集版本或实验环境的不同设置来改变实验结果。我们建议该模型的潜在用户通过交叉验证来确定他们自己的最佳超参数。
 # 5. 结论 在本研究中,我们提出了一种新的深度学习模型,以更准确地预测DDIs的药理作用。使用每种药物的SSP、TSP和GSP三个相似度轮廓来训练所提出的模型。使用自动编码器来减少这些相似性,并将其输入一个深度前馈网络来预测每个DDI的类型。与现有模型相比,该模型的分类精度得到了提高。我们发现GSP和TSP可以提高预测性能。我们还预测了许多ddi的新效应,其中许多都得到了许多数据库或以前的研究的支持。
# 5. 结论 在本研究中,我们提出了一种新的深度学习模型,以更准确地预测DDIs的药理作用。使用每种药物的SSP、TSP和GSP三个相似度轮廓来训练所提出的模型。使用自动编码器来减少这些相似性,并将其输入一个深度前馈网络来预测每个DDI的类型。与现有模型相比,该模型的分类精度得到了提高。我们发现GSP和TSP可以提高预测性能。我们还预测了许多ddi的新效应,其中许多都得到了许多数据库或以前的研究的支持。
6. 方法
我们使用了三种相似性度量,分别是结构相似性描述(SSP)、目标基因相似性描述(TSP)和基因本体(GO)术语相似性描述(GSP)。
药物A的SSP是A与其他药物之间结构相似值的向量。两种药物之间的结构相似性是由它们的smiles转换成它们的二进制向量(指纹)之间的Tanimoto系数。药物A的SSP可以表示为SSPA = {SSAA, SSAB, SSAC,…},其中SSAx为药物A 与X之间的Tanimoto系数。
药物A的TSP是A与其他药物靶基因相似值的载体。药物A与药物B的靶基因相似性计算公式如下: 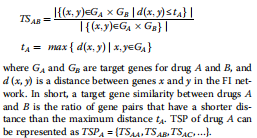 GSP计算方法与TSP计算方法相同,只是将基因和FI网络分别替换为GO项和GO图。药物A的GSP可以表示为GSPA = {GSAA, GSAB, GSAC,…},其中GSAB与TSAB相似。一种药物的SSP、TSP和GSP的长度为1597,与所有药物的数量相同。
GSP计算方法与TSP计算方法相同,只是将基因和FI网络分别替换为GO项和GO图。药物A的GSP可以表示为GSPA = {GSAA, GSAB, GSAC,…},其中GSAB与TSAB相似。一种药物的SSP、TSP和GSP的长度为1597,与所有药物的数量相同。
本研究从DrugBank获取DDI数据,运用NLP技术根据描述语法将DDI相关事件划分为65种类型,编译了包含572种药物、74 528种交互和65种DDI相关事件的数据集。提出了一种基于深度学习的多模式深度学习框架DDIMDL,该框架将多种药物特性与深度学习相结合,用于DDI事件预测。使用5-CV评估,DDIMDL优于现有的DDI事件预测方法和基线方法。综上所述,语义分析的使用使我们能够对DrugBank事件进行显著的分类,而多模式学习为整合不同特征和花费合理的训练时间提供了有力的途径。多模态深度学习框架是一种很有前途的DDI事件预测工具。
 预测DDI类型的模型
预测DDI类型的模型
DDI类型的预测模型由三个自编码器和一个深度前馈网络组成。采用自动编码器对SSP、TSP和GSP进行降维。3个自动编码器都是同构的,输入层和输出层大小分别为3194(= 1597×2)和600,隐含层大小分别为1000、200和1000。深度前馈网络具有大小为600的输入层,6个大小为2000的隐藏层和大小为106的输出层。
batch大小为256,自编码器学习率为0.001,前馈网络学习率为0.0001。自动编码器和前馈网络的激活函数为sigmoid和ReLU。我们使用sigmoid作为前馈网络输出层的激活函数。epoch的数量是850,使用Adam作为前馈网络的优化器,使用RMSprop作为autoencoder优化器。为了避免过拟合,我们对前馈网络和autoencoders应用了drop rate为0.3的dropout和批处理归一化。
对于每个epoch,分别训练三个自动编码器,使输入输出的差异最小化。训练的目的是使三个自动编码器和前馈网络的总代价最小化。