通过整合药物表型、治疗性、化学和基因组特性,基于机器学习的药物-药物相互作用预测
0. 摘要
目的:药物-药物相互作用(drug - drug interaction, dis)是药物开发和临床应用的重要考虑因素,尤其是联合用药。虽然在临床试验期间有必要确定所有可能的DDIs,但DDIs经常是在药物被批准临床使用后报告的,它们是引起药物不良反应(ADR)和增加医疗保健成本的常见原因。计算方法可以在临床试验中帮助识别潜在的DDIs。
方法:提出一种异构网络辅助推理(HNAI)框架来辅助预测DDIs。首先,我们基于DrugBank数据构建了一个完整的DDI网络,该网络包含721个获批药物的6946对DDI。接下来,我们分别计算药物四个特征之间的相似性:药物表型、治疗效果、化学和基因组特性。最后,我们在HNAI框架中应用了五种模型来预测DDI:朴素贝叶斯、决策树、k近邻、logistic回归和支持向量机。
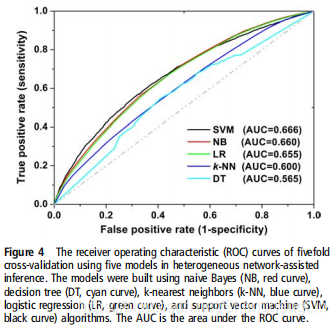
结果:通过五折交叉验证,HNAI模型的ROC曲线下面积为0.67。以抗精神病药物为例,一些预测的DDIs得到了文献的支持。
结论:基于机器学习的药物表型、治疗、结构和基因组相似性的整合,我们证明了HNAI在药物开发和上市后监测中发现DDIs是有希望的。 # 1. 介绍
药物-药物相互作用(DDIs)发生在联合用药过程中。它们是药物不良反应(ADRs)的常见原因,并导致医疗保健成本增加。许多DDIs在临床试验阶段没有被发现,在药物被批准临床使用后才被报道。此类DDIs往往导致患者的发病率和死亡率,占所有住院患者用药错误的3-5%。临床DDIs也会导致严重的社会和经济问题。因此,在药物被批准或使用之前,迫切需要检测或确定DDIs。
目前,DDI预测侧重于检测代谢谱,例如细胞色素P450 (CYP450) 或转运体相关的药代动力学相互作用。然而,利用实验方法识别DDI的能力有限是药物开发过程中的主要障碍。在过去十年中,设计并提供了几种方法来预测潜在的DDI。
在本研究中,我们提出了一种异质网络辅助推理(HNAI)框架(图1),用于大规模预测可能发生的DDIs。首先,我们构建了一个完整的DDI网络,该网络包含6946对高质量的DDI对,连接了DrugBank数据库中721种获批药品。接下来,我们计算了四种类型的药物-药物相似性作为每个药物-药物对的特征。其中三种药物相似性(表型相似性、治疗相似性和结构相似性)用先前的方法计算。此外,我们基于DrugBank和治疗靶标数据库(Therapeutic Target Database, TTD)构建的大药物-靶标相互作用(DTI)网络引入了一种新型药物特征,即药物的基因组相似性。最后,我们在HNAI框架中应用了五种机器学习算法作为预测模型:朴素贝叶斯(NB)、决策树(DT)、k近邻(k-NN)、logistic回归(LR)和支持向量机(SVM)。通过五折交叉验证,我们证明了HNAI获得了高性能。在我们之前的工作中,我们已经展示了药物表型、治疗和结构相似性的整合对DTI预测的潜在价值。在这里,我们扩展了这种方法,利用基于机器学习的药物表型、治疗、结构和基因组相似性的整合来预测DDI。与之前的工作相比,本研究有三个改进之处:(i)我们引入了另外一个重要的药物相似类型(基因组相似度),并通过将药物基因组相似度和先前报道的三种药物相似度相结合来建立一个框架用于DDI预测;(ii)系统评估了五种机器学习算法,并在HNAI框架中建立了预测模型;(iii)虽然我们使用现有的网络构建方法来计算药物表型相似度,但之前的药物不良反应网络仅使用MetaADEDB的数据来构建。在本研究中,我们通过整合MetaADEDB和美国食品药品监督管理局(US FDA)创建的FDA不良事件报告系统(FAERS)的数据,建立了一个更全面的药品不良反应网络。总的来说,我们的工作可以提供一种替代工具,并可能在药物开发和上市后监测中改善配体受体DDI预测。 # 2. 方法
2.1 数据收集
DDI网络
我们从DrugBank数据库收集DDI数据(V.3.0;http://www.DrugBank.ca/)。在批准和实验药物中有超过16453对DDI。这些DDIs可分为三种类型:药物的相互作用、药代动力学相互作用和药效学相互作用。我们删除了不能用于计算四类相似度的药物,并且删除了不具有副作用信息的药物。因此,我们专注于预测可能发生在先前确定的配体-受体位点的药代动力学和药效学相互作用(配体-受体DDIs)。我们总共收集了6946个高质量、独特的配体-受体DDI对,连接了721个获批药物,用于模型构建和验证。我们从DrugBank数据库中检索药品的SMILES数据,然后使用Open Babel29 (V.2.3.1)将其转换为规范的SMILES。
药物不良反应关联及其网络(Drug–ADR associations and their network)
我们准备了两个数据集。第一个药物-ADR数据集是从最近发布的数据库MetaADEDB下载的。第二个数据集是从FAERS获得的。最后,我们利用先前的方法计算药物表型相似性,通过去除FAERS和MetaADEDB之间重复的药物不良反应相关性,获得了唯一的药物不良反应相关性。
药物靶标相互作用及其网络
我们从DrugBank和TTD两个数据库收集DTI数据。我们总共获得了2912对连接674个独特的靶蛋白和721个获批药物的DTI。我们根据DrugBank数据库中的ATC代码标注了所有药物的详细治疗信息。
2.2 四种相似性的度量
表型相似性Sp(di,dj),治疗相似性St(di,dj),化学结构相似性Ss(di,dj)是根据之前的工作计算的。此外,我们引入了基因组相似性Sj(di,dj)来描述本研究中的一个药物-药物对。由于基因组相似性,每种药物都使用靶蛋白位向量编码(图1C)。每个向量元素代表一个靶标蛋白。如果靶标蛋白在DTI网络中与药物相关联,则相应的位设为' 1 ',否则设为' 0 '。然后,药物i和药物j的相似性通过计算药物的靶蛋白位向量的Tanimoto系数得到。
2.3 利用HNAI开发预测模型
图1展示了HNAI的整个计算框架。我们收集了6946个高质量、独特的药物-药物对作为阳性的DDI集合。这里阳性的DDI对是临床报道的DDI。然后,我们从721个批准的药物中随机选择了相同数量的非DDI对(6946对)作为阴性的DDI集合。每种药物-药物对用四种不同的相似性来表示:Sp(di,dj),St(di,dj),Ss(di,dj),Sj(di,dj)。最后,我们使用五种机器学习算法算法,分别为NB、DT、k-NN、LR和SVM,在HNAI上实现了预测模型。其中四种算法(NB, DT, k-NN和LR)在Orange Canvas (V.2 .0b; http://www.ailab.si/orange/)上实现。我们使用LIBSVM包(V.3.1)构建了SVM模型。
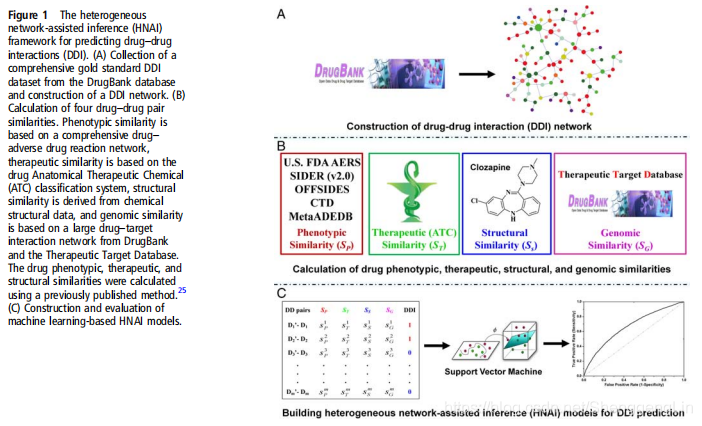 ## 2.4 HNAI模型的评估
## 2.4 HNAI模型的评估
在这项研究中,我们使用五折交叉验证技术来评估所有模型的性能。使用ROC曲线来评估每个模型的性能。
3. 结果
已知DDI网络的构建
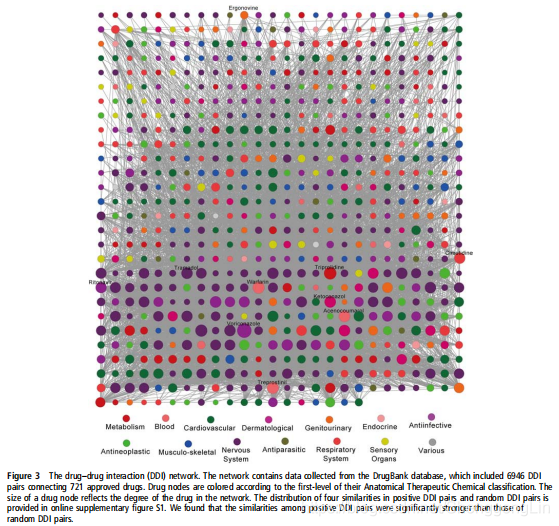
我们从DrugBank数据库收集了一个大型配体受体DDI数据集。本数据集包括6946个DDI对,连接721个美国fda批准的小分子药物。在图2中,药物按一级ATC代码分组。我们发现基于一级ATC代码,大多数DDIs发生在不止一类药物中。例如,我们在DDI网络中发现了487种度大于5的药物。总共,487种药物中有106种属于心血管系统。此外,173种神经系统药物(紫色)的平均度为25.8,显著高于548种非神经系统药物的17.2。总的来说,心血管系统和神经系统药物是DDIs的高风险药物。 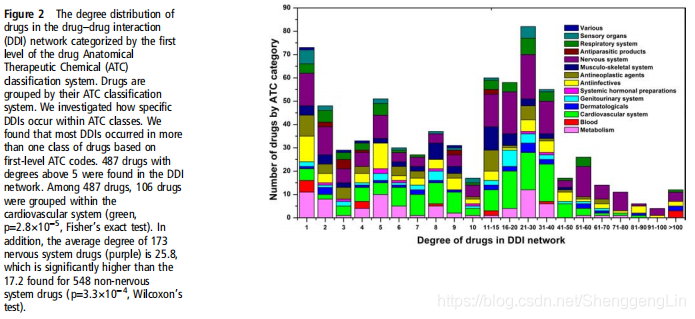 图3使用Cytoscape的网格布局以图形方式表示DDI网络。它包括6946对高质量DDI连接721个批准的药物。在图3中,一个节点表示一种药物,一个边表示两种有临床报道或文献管理的DDIs药物之间的关联。每个节点都根据一级ATC编码进行颜色编码。网络中平均药物度为19.3,度分布为幂律分布(y¼axb, a=94.0,
b=−0.19)。
图3使用Cytoscape的网格布局以图形方式表示DDI网络。它包括6946对高质量DDI连接721个批准的药物。在图3中,一个节点表示一种药物,一个边表示两种有临床报道或文献管理的DDIs药物之间的关联。每个节点都根据一级ATC编码进行颜色编码。网络中平均药物度为19.3,度分布为幂律分布(y¼axb, a=94.0,
b=−0.19)。
4. HNAI模型的表现
 # 5. 模型限制和未来工作
# 5. 模型限制和未来工作
HNAI框架存在若干限制。首先,它缺乏一个“黄金标准”的非DDI数据集作为阴性的DDI来构建模型。我们不能确定两种药物在非DDI数据集中不相互作用。其次,四种药物相似性的准确性是有限的。为了评估整个HNAI 模型中每个相似度的相对重要性,我们利用三种不同相似度的组合来重建机器学习模型。我们发现去掉结构或表型特征后,模型的性能较差。因此,与其他两个特征相比,结构和表型特征对模型的性能是重要的。第三,数据不完整是一种限制。尽管我们使用了两个大型网络:一个药物不良反应关联网络和一个DTI网络,来测量药物表型和基因组相似性,但两个网络都是不完整的。最后,每一种相似性都可能有几个附加的限制。虽然利用MACCS键计算出的化学二维(2D)结构相似性已成功用于DDI或ADR评估,但6 20个二维结构相似的分子在三维结构中可能有非常不同的形状。此外,二维结构上不同的化合物在三维结构上可能有非常相似的形状,这可能在配体-受体结合中发挥关键作用。在这里,利用来自DrugBank和TTD的广泛DTI数据计算基因组相似性。然而,由于缺乏作用模式、生化作用和结合位置的数据,详细的药物靶标信息在目前的HNAI框架中丢失了。
然而,我们使用HNAI框架预测了数千个可能的DDI。未来,我们计划利用Vanderbilt公司的合成衍生(SD)数据库(https://starbrite.vanderbilt.edu/biovu/sddata.html)验证更多预测的DDI。我们还打算:(i)通过从Vanderbilt’s SD数据库中整合更有用的药物3D形状相似性特征、全面的DTI网络和ehr来改进HNAI框架;(ii)通过整合Vanderbilt DNA库的药物反应数据和全基因组基因分型数据,探讨DDIs的分子机制和遗传图谱(https://starbrite.vanderbilt.edu/biovu/)。
6. 结论
通过利用药物表型、治疗性、结构和基因组相似性,我们提出了一种预测DDIs的HNAI框架。我们在来自DrugBank数据库的大型DDI数据集上应用了五种基于机器学习的预测模型。基于五折交叉验证,SVM模型的AUC值为0.67。在我们对几种涉及抗精神病药物的新型预测DDIs的探索中,我们论证了HNAI框架在识别抗精神病药物DDIs方面的潜在效用。总之,我们证明了基于机器学习的药物表型、治疗、结构和基因组相似性的整合使用系统药理学方法是一种简单而有效的预测未知DDI的策略。
原论文名称:Machine learning-based prediction of drug–drug interactions by integrating drug phenotypic, therapeutic, chemical, and genomic properties