使用机器学习来测量基因间的相关性一个多特征模型
使用机器学习来测量基因间的相关性:一个多特征模型
1. 摘要
测量一对基因间的条件亲缘关系是计算生物学的一项基本技术,也是一个重大的挑战。论文提出了一个新的机器学习模型—多特征相关性(MFR),通过将表达相似度和基于先验知识的相似度纳入评估标准,来准确地测量一对基因之间的条件相关性。
2. 介绍
基因之间的相互作用通常被建模为一对基因之间0/1(非相互作用/相互作用)的二元关系,而亲缘性则意味着一对基因之间的某种程度的关系。
相关性可以通过两种特征来衡量:表达相似度和基于先验知识的相似度。第一种特性通常是在一定条件下测量一对基因的共表达水平,第二种类型的特征通常是使用公共生物数据和功能注释数据库来测量基因的相关性。
论文提出的机器学习模型MFR,通过使用带线性核的支持向量机,整合表达相似度和基于先验知识的相似度,保留并推荐具有高表达相似性和高先验知识相似性的基因对,准确地测量基因间的条件相关性。
3. 材料和方法
3.1 MFR工作流程
如图1所示,MFR工作流程有5个步骤: (i) 从已发表的研究成果中收集基因对样本; (ii) 从GEO、GO和orthoDB数据库中提取基因特征,用于评估基于相似性的基因对功能; (iii) 利用4个基因特征和反应体数据库和HTRIdb数据库计算12个基于相似性的基因对特征 (iv) 通过10倍交叉验证构建基于svm的模型。 (v)实验验证基因-基因相互作用,预测基因功能,并与其他模型和方法进行比较。 ## 3.2 基因特征 MFR使用12个相似的基因对特征来评估一对基因之间的条件相关性。 表达数据。使用GEO数据库中的15679个样本作为表达数据源,进行预处理步骤,最终保留16,391个编码蛋白的基因,以供进一步的表达数据分析。 基因本体论数据。 GO注释使用GO数据库(共435975)中43340个与实验相关的生物学过程。 相应的数据。使用了5000多个物种的2200万个基因,其中包括20个物种的169376个人类同源基因。
亚细胞定位数据。从GO数据库中获得的人类基因的160537个细胞成分注释被用作亚细胞源来衡量一对基因之间亚细胞定位的相似性。 ## 3.3 基因对特征计算 MFR中使用了12个基于相似性的基因对特征,定义如下。 7个基于表达相似性的特征:每个基因exp1和exp2的平均表达水平,PCC度量的线性共表达关系,SRC和MI用于测量的非线性共表达关系,MI度量的两个基因表达水平的联合分布与因子边际分布的产物相似性。 GO相似性(goSim),因为相互作用的基因被认为参与了类似的生物过程: 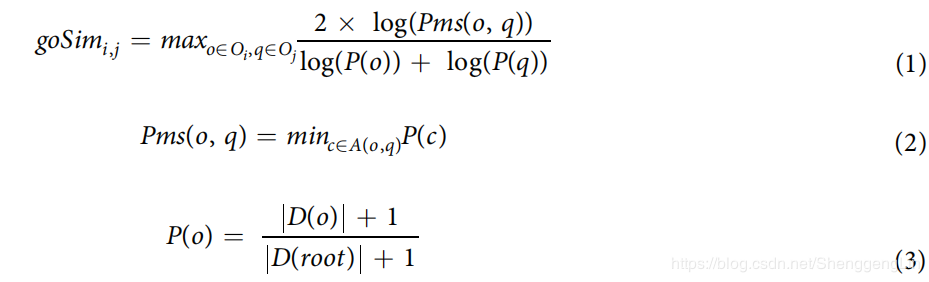 其中,Oi和Oj分别表示用于注释基因i和j的GO项集;A(o, q)是GO项o和q的共同祖先集;P(o)为GO项o实例注释的一个基因的概率;D(o)和D(root)分别表示GO项o的后代GO项集和根GO项集。 亚细胞定位相似度(lcSim),用来计算两个蛋白质编码基因出现在一个普通细胞器中的概率:
其中,Oi和Oj分别表示用于注释基因i和j的GO项集;A(o, q)是GO项o和q的共同祖先集;P(o)为GO项o实例注释的一个基因的概率;D(o)和D(root)分别表示GO项o的后代GO项集和根GO项集。 亚细胞定位相似度(lcSim),用来计算两个蛋白质编码基因出现在一个普通细胞器中的概率:
 其中Li和Lj是由基因i和j编码的亚细胞定位组。 同源相似性(hgSim),使用改进的皮尔逊相关方法:
其中Li和Lj是由基因i和j编码的亚细胞定位组。 同源相似性(hgSim),使用改进的皮尔逊相关方法: 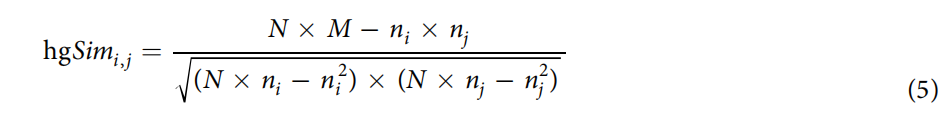 其中,ni和nj分别为基因组中包含i和j基因同源基因的物种数目;N =21 是我们使用的物种 的总数和,M为基因组中同时包含i和j基因同源基因的物种数量。 基因对的归一化距离(rxSim),从反应体途径得到的202772个基因-基因相互作用被用来构建一个未加权图,其中节点表示基因,边表示基因之间的相互作用:
其中,ni和nj分别为基因组中包含i和j基因同源基因的物种数目;N =21 是我们使用的物种 的总数和,M为基因组中同时包含i和j基因同源基因的物种数量。 基因对的归一化距离(rxSim),从反应体途径得到的202772个基因-基因相互作用被用来构建一个未加权图,其中节点表示基因,边表示基因之间的相互作用: 
其中disi,j为基因i与j之间的最短距离,dismax为图中最远的一对基因之间的最短距离。 基于转录调控相似度(trSim),如果有一个基因对有转录调控相互作用的记录,trSim为1,否则为0。
3.4 支持向量机模型
基于支持向量机设计MFR,并结合相关的学习算法进行分类和回归分析。取12个基于相似性的基因对特征作为输入,输出值作为评价标准,用于检测一对基因之间的条件相关性(见下图)。 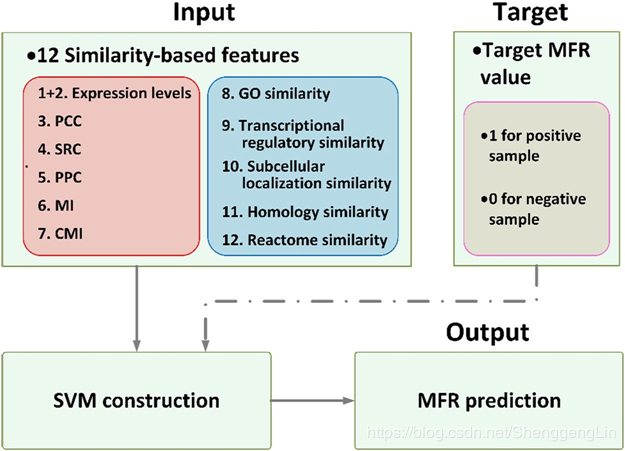 使用提供标签MFR值(标记为1/0--阳性/阴性)的基因对用于模型训练。给定X ={x1, x2,..., xn} 和 Y= {y1, y2,..., yn}, 式中,xi和yi表示12个基于相似性的基因对特征向量和第i个基因对的目标MFR值(label),建立MFR模型如下:
使用提供标签MFR值(标记为1/0--阳性/阴性)的基因对用于模型训练。给定X ={x1, x2,..., xn} 和 Y= {y1, y2,..., yn}, 式中,xi和yi表示12个基于相似性的基因对特征向量和第i个基因对的目标MFR值(label),建立MFR模型如下: 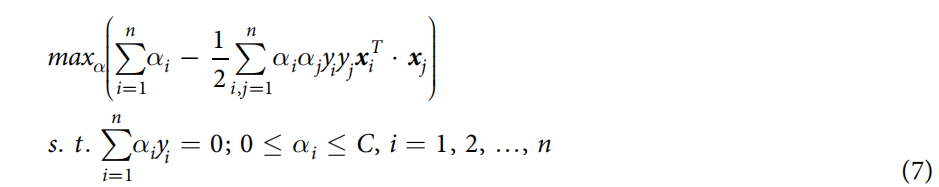 式中,α={α1,α2, ...,αn}表示拉格朗日乘数,采用序列最小优化方法求解。预测第i个基因对的
MFRi值被定义为:
式中,α={α1,α2, ...,αn}表示拉格朗日乘数,采用序列最小优化方法求解。预测第i个基因对的
MFRi值被定义为: 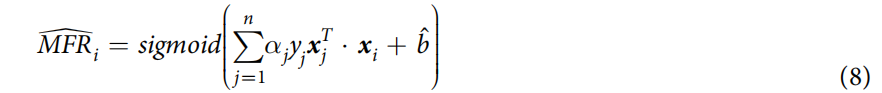 由于高表达相似度和基于先验知识的相似度的阳性基因对不够直接用于训练,收集高表达相似度的阳性基因对和相应的阴性基因对组成共表达子数据集。同样,收集具有高优先级知识相似度的阳性基因对和相应的阴性基因对,组成优先级知识库子数据集。MFR的训练数据集包括这两个数据集。MFR值越高,说明两个基因更有可能相互作用。 # 4. 结果 对MFR模型进行了10次交叉验证,再使用MFR模型进行一个测试验证和两个进一步的验证,最后,利用MFR模型构建肿瘤基因网络,预测基因功能。所有结果与其他模型或方法进行比较(见下表)。
由于高表达相似度和基于先验知识的相似度的阳性基因对不够直接用于训练,收集高表达相似度的阳性基因对和相应的阴性基因对组成共表达子数据集。同样,收集具有高优先级知识相似度的阳性基因对和相应的阴性基因对,组成优先级知识库子数据集。MFR的训练数据集包括这两个数据集。MFR值越高,说明两个基因更有可能相互作用。 # 4. 结果 对MFR模型进行了10次交叉验证,再使用MFR模型进行一个测试验证和两个进一步的验证,最后,利用MFR模型构建肿瘤基因网络,预测基因功能。所有结果与其他模型或方法进行比较(见下表)。 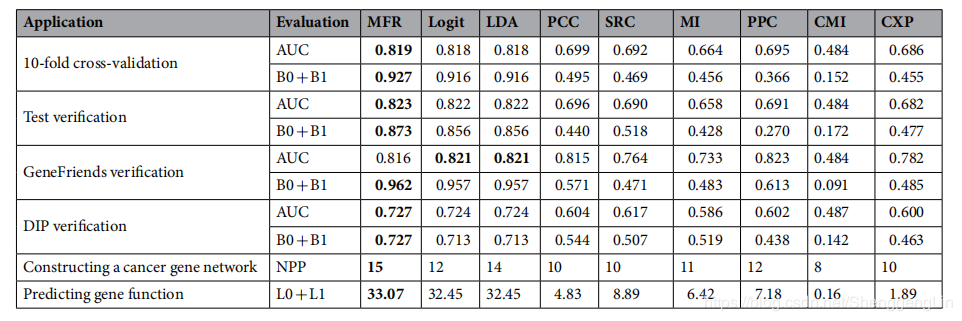 结果表明,MFR在识别基因-基因相互作用的AUC值最高。具体来说,与其他线性模型和共表达分析方法相比,该方法在所有数据集中检测高表达相似性和基于先验知识相似性的基因对的准确率平均提高了1.1%。在肿瘤基因网络构建和基因功能预测方面,MFR也比其他模型和方法获得了更有生物学意义和平均预测精度更高的结果。
结果表明,MFR在识别基因-基因相互作用的AUC值最高。具体来说,与其他线性模型和共表达分析方法相比,该方法在所有数据集中检测高表达相似性和基于先验知识相似性的基因对的准确率平均提高了1.1%。在肿瘤基因网络构建和基因功能预测方面,MFR也比其他模型和方法获得了更有生物学意义和平均预测精度更高的结果。