DeepPurpose a deep learning library for drug–target interaction prediction
本篇推文引自:DeepPurpose: a deep learning library for drug–target interaction prediction
1. 摘要
准确预测药物靶点相互作用(DTI)是药物发现的关键。近年来,深度学习(DL)模型在DTI预测方面表现出了良好的性能。然而,这些模型对于进入生物医学领域的计算机科学家和缺乏DL经验的生物信息学家来说都很难使用。我们提出了DeepPurpose,一个全面且易于使用的DTI预测DL库。DeepPurpose通过实现15个化合物和蛋白质编码器以及50多个神经结构来支持定制的DTI预测模型的训练,同时还提供了许多其他有用的特性。我们演示了DeepPurpose在几个基准数据集上的最新性能。
2. 介绍
药物-靶标相互作用(DTI)用于描述化合物与蛋白靶标的结合(Santos等,2017)。准确识别药物分子靶点是药物发现和开发的基础(Rutkowska et al., 2016;Zitnik等人,2019年),并且对于有效和安全的治疗新病原体也很重要,包括SARS-CoV-2 (Velavan和Meyer, 2020年)。
深度学习(DL)在识别、处理和推断分子数据中的复杂模式方面提供了增强的表达能力,从而提高了化合物的传统计算建模(Lee等人,2019年;O¨ztu¨rk等人,2018年)。有许多用于DTI预测的DL模型(Lee等人,2019;Nguyen等人,2020年;O¨ztu¨rk等人,2018年)。然而,要生成预测、在实践中部署DL模型、测试和评估模型性能,需要相当多的编程技能和广泛的生化知识。流行的工具是为有经验的跨学科研究人员设计的。它们对进入生物医学领域的计算机科学家和在培训和部署DL模型方面经验有限的领域生物信息学家来说都具有挑战性。此外,每个开源工具都有不同的编程接口,而且编码也不同,这就阻碍了对模型集成的各种方法的输出的简单集成(Yang et al., 2019)。
在这里,我们介绍DeepPurpose,一个用于蛋白质和化合物编码和下游预测的DL库。DeepPurpose允许通过编程框架实现超过50个DL模型,7个蛋白质编码器和8个复合编码器快速原型。经验上,我们发现采用DeepPurpose的模型在DTI基准数据集上实现了最先进的预测性能。
3. DeepPurpose库
用于DTI预测的DL模型可以表述为编解码器架构(Cho等,2014)。DeepPurpose库实现了统一的编码器-解码器框架,这使得该库具有独特的灵活性。只需指定编码器的名称,用户就可以自动地将感兴趣的编码器与相关的解码器连接起来。然后DeepPurpose以端到端的方式训练相应的编码器-解码器模型。最后,用户通过编程或可视化界面访问训练好的模型,并使用该模型进行DTI预测。
3.1 编码蛋白质和化合物的模块
DeepPurpose以化合物的简化分子输入系统(SMILES)串和蛋白质氨基酸序列对为输入。然后,它们被输入分子编码器,该编码器指定了一种深度转换功能,将化合物和蛋白质映射到一个矢量表示。特别地,对于化合物,DeepPurpose提供了八种使用化合物不同模式的编码器:Multi-Layer Perceptrons (MLP) on Morgan, PubChem,Daylight and RDKit 2D Fingerprint; Convolutional Neural Network (CNN) on SMILES strings;Recurrent Neural Network (RNN) on top of CNN; transformer encoders on substructure fingerprints; message passing graph neural network on molecular graph.对于蛋白质,DeepPurpose为输入的氨基酸序列提供了7个编码器:MLP on
Amino Acid Composition (AAC), Pseudo AAC, Conjoint Triad, Quasi-Sequence descriptors; CNN on amino acid sequences; RNN on top of CNN; transformer encoder on substructure fingerprints.注意,可选的输入特性可能不适用于特定的编码器体系结构。详细的编码器规格和参考资料在补充材料中被描述。 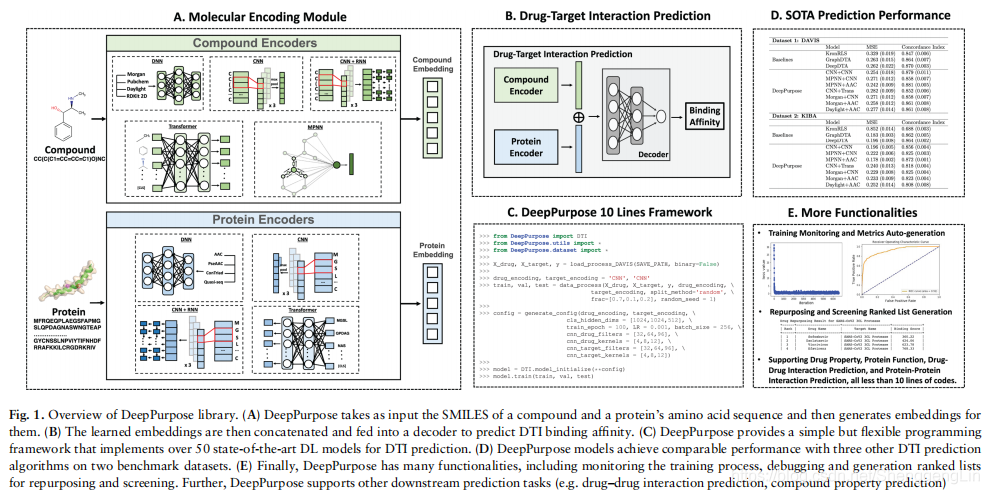
3.2 DTI预测模块
DeepPurpose将学到的蛋白质和化合物嵌入到MLP解码器中以产生预测。输出分数包括连续结合分数,如中位抑制浓度(IC50),以及表示蛋白是否与化合物结合的二值输出。库检测任务是回归还是分类,并切换到正确的丢失函数和评估指标。在回归的情况下,我们使用均方误差(MSE)作为损失函数,并使用均方误差、一致性指数和皮尔逊相关作为绩效指标。在分类案例中,我们采用二元交叉熵作为损失函数,以接收机工作特性下的面积(AUROC)、精确召回下的面积(AUPRC)和F-1评分作为性能指标。在推理时,给定新的蛋白质和化合物,DeepPurpose返回预测分数,表示化合物和蛋白质结合的预测概率。
3.3 其他下游预测任务模块
DeepPurpose包括repurpose和virtual_screening函数。只用几行代码指定要筛选化合物库的列表和一组可选的训练数据集,DeepPurpose训练五个DL模型,综合预测结果并生成一个可描述的排名最高的化合物。如果用户没有指定训练数据集,DeepPurpose将使用预先训练好的深度模型进行预测。然后可以对这个列表进行检查,以确定有希望的化合物候选物,供进一步的实验使用。其次,DeepPurpose还为其他建模任务支持用户友好的编程框架,包括药物和蛋白质特性预测、药物-药物相互作用预测和蛋白质-蛋白质相互作用预测(见补充材料)。第三,DeepPurpose提供了许多类型数据的接口,包括公共大型绑定亲和力数据集(Liu et al.,2007)、生物测定数据(Kim et al., 2019)和药物再利用库(Corsello et al., 2017)。
3.4 编程框架和实现细节
DeepPurpose的功能被模块化为6个关键步骤,其中一行代码可以调用每一个步骤:(i)从本地文件加载数据集,或者加载DeepPurpose基准数据集。(ii)指明化合物和蛋白质编码器的名称。(iii)使用data_process函数将数据集拆分为训练集、验证集和测试集,实现了多种数据拆分策略。(iv)创建一个配置文件,并指定模型参数。如果需要,DeepPurpose可以自动搜索超参数的最优值。(v)使用配置文件初始化模型。或者,用户可以加载一个预先训练好的模型或先前保存的模型。(vi)最后,利用train函数对模型进行培训,监控培训进度和绩效指标。DeepPurpose与操作系统无关,它使用Jupyter笔记本接口。它可以运行在云或本地。所有数据集、模型、文档、安装说明和教程都提供在https://github.com/kexinhuang12345/DeepPurpose。
4. 使用DeepPurpose 用于 DTI prediction
为了演示DeepPurpose的使用,我们将DeepPurpose与KronRLS (Pahikkala et al., 2015),一种流行的DTI方法,以及GraphDTA (Nguyen et al., 2020)和DeepDTA (O¨ztu¨rk et al.,2018),最先进的DL方法进行比较。我们发现,许多DeepPurpose模型在两个基准数据集——DAVIS (DAVIS et al., 2011)和KIBA(He et al., 2017)上的预测性能相当(图1D)。补充材料中提供了生成结果的完整脚本。
5. 具有交互式的web界面
除了快速的模型原型,DeepPurpose还提供了实用函数来加载预先训练好的模型,并对新药和目标输入进行预测。该功能允许领域科学家快速检查预测,根据预测修改输入,并在此过程中迭代,直到找到具有所需属性的药物或目标。我们利用Gradio (Abid等人,2019)以编程方式创建web界面。我们在后端使用一个用户训练过的DeepPurpose模型,并用不到10行代码创建一个定制的web界面。这个web界面以smile和氨基酸序列作为输入,并以小于1秒的延迟返回预测分数。我们在补充材料中提供了例子。