A cancer drug atlas enables synergistic targeting of independent drug vulnerabilities
本篇推文引自:A cancer drug atlas enables synergistic targeting of independent drug vulnerabilities
1. 摘要
使用联合药物协同作用进行个性化的癌症治疗是有吸引力的,但同时也是一件困难的事情。在这里,我们提出了一种方法来揭示基于单药效应分析的药物组合的有效性。为此,我们使用了来自药物基因组百科全书的剂量反应数据,并将这些数据表示为药物图谱。药物图谱代表了药物作用之间的关系,并允许识别肿瘤在被两种药物攻击时的独立过程。我们的方法能够预测与肿瘤驱动突变有关的联合治疗。通过使用这一策略,我们可以在泛癌症范围内发现潜在的有效药物组合。预测的协同作用已经在胶质母细胞瘤、乳腺癌、黑色素瘤和白血病小鼠模型中得到验证,在75%的测试模型中产生了治疗协同作用。这表明我们可以准确预测有效的药物组合,具有临床转化价值。
2. 结果
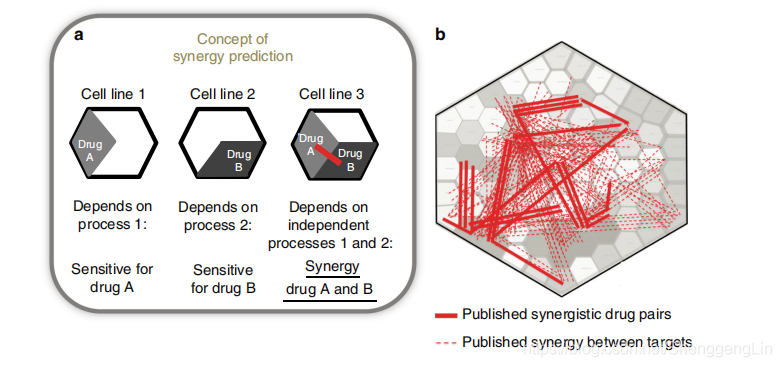
一种药物图谱允许可视化复杂的协同药物相互作用。现有的抗癌药可能有效的组合是巨大的,需要一种合理的方法来选择最有潜力的组合,同时也考虑到每个病例的基因背景。我们推断癌症过程之间的关系可以通过药物作用之间的关系来反映。因此,药物反应数据可能会指导我们开发同时影响肿瘤驱动过程的联合疗法。图1a举例说明了我们的方法:如果一个细胞系对药物a敏感而对药物B不敏感,或者相反,那么潜在的过程显然是独立工作的。然而,如果第三个细胞系同时对药物a和B敏感,那么这些独立的过程就可以同时靶向,形成共同的易感性。我们认为,我们可以利用这一概念来识别癌细胞系中常见的共脆弱性,并且当这些细胞使用正确的联合疗法治疗时,可以预期会有更多的附加(协同)药物效应。
为了确定哪些过程是独立工作的,我们计算了多个细胞系中单一药物作用的差异水平。对许多细胞系有相似反应的药物最终会聚集在同一个簇中,而对许多细胞系有不同反应的药物聚集在相对更远的簇中。为了可视化这些聚类距离关系,聚类树被投影为一个2D Voronoi图,我们称之为药物图谱。这个药物图谱提供了对许多细胞系上的药物效应关系的直观概述。利用世界地图坐标关系并行验证了atlas方法(补充图1a,另见补充图1b-d)。
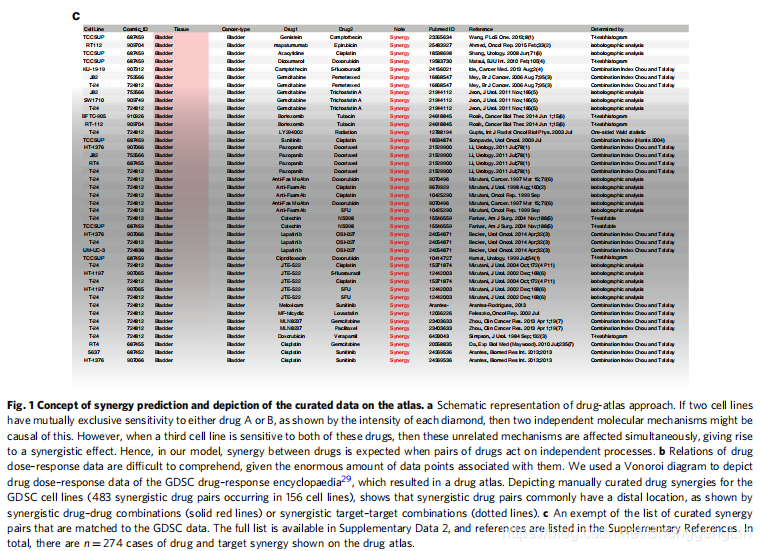
为了研究具有协同效应的药物组合如何与药物效应相似性相关,我们整理了所有发表的和同行评阅的协同效应数据,这些数据与用于创建药物图谱的细胞系数据相匹配。结果共鉴定出483对药物在156个细胞系中表现出协同作用(补充数据2,参考文献见补充文献)。这些协同作用通过在各自的药物之间画一条线显示在药物图谱上(图1b,更详细的药物靶点视图22在补充图1e中给出)。图1c显示了一组经过整理的数据。
 与我们的概念一致,大多数协同药物相互作用在图谱上跨越很大的距离,以定性的方式显示相应的药物对影响不相关的过程。
与我们的概念一致,大多数协同药物相互作用在图谱上跨越很大的距离,以定性的方式显示相应的药物对影响不相关的过程。
药物距离、药物敏感性和靶向治疗与药物协同作用相关。基于我们的概念,我们期望抑制不相关的过程将导致协同效应,因为它们代表独立的生存机制。为了量化这一点,我们使用药物距离计算了药物效应的不同程度。药物效应已经聚类生成药物图谱,并用于计算聚类之间的遗传距离。同源距离越大,药物效应越不相关,药物距离越高。具有协同效应的药物对的平均药物距离显著超过了平均总药物距离图2a, P = 4×10−4),证实了我们的初始假设。其他距离模型(平均聚类)显示了类似的结果(见补充图2a)。同样地,我们确定药物距离,我们可以计算靶标距离,因为每个药物都有一个确定的靶标。
由于我们发现了GDSC数据中存在的细胞系的协同药物对,我们能够将药物敏感性数据与在特定情况下显示协同作用的细胞系进行匹配。从我们的模型中,我们期望在发生协同作用时具有更高的敏感性。因此,我们测试了协同作用的发生是否与药物敏感性相关。该分析确实表明,与匹配肿瘤类型的对照细胞株相比,与特定药物协同作用的细胞株对这些药物明显更敏感(图2e, Poverall = 1×10−4,Supplementary Fig. 2d, P = 6×10−4)。
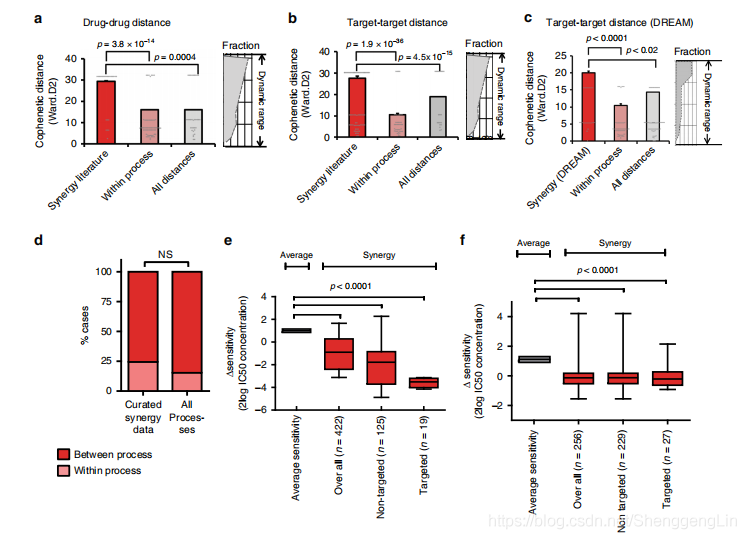
 分析表明,药物距离与相应药物之间发生协同作用之间存在明显的正相关关系。此外,具有协同作用的细胞系通常对这些药物相对敏感,特别是在靶向肿瘤驱动突变的情况下。我们能够生成协同作用发生的预测模型,该模型表现出良好的性能。一些体内实验的结果也证实了我们的协同预测模型在五个独立的小鼠模型中的有效性,并表明该预测模型具有临床转化价值。 # 3. 方法 细胞系的分子特征和药物靶点。本研究中使用的数据集在补充数据1中描述,并参考了Sanger GDSC癌细胞系(以及Novartis/Broad CCLE数据)。对于表达分析,使用了Cosmic财团的数据(使用Affymetrix HT-HG-u133av2平台对789个癌细胞系进行基因表达分析,来源:EBI ID:E-MTAB-78322)。
分析表明,药物距离与相应药物之间发生协同作用之间存在明显的正相关关系。此外,具有协同作用的细胞系通常对这些药物相对敏感,特别是在靶向肿瘤驱动突变的情况下。我们能够生成协同作用发生的预测模型,该模型表现出良好的性能。一些体内实验的结果也证实了我们的协同预测模型在五个独立的小鼠模型中的有效性,并表明该预测模型具有临床转化价值。 # 3. 方法 细胞系的分子特征和药物靶点。本研究中使用的数据集在补充数据1中描述,并参考了Sanger GDSC癌细胞系(以及Novartis/Broad CCLE数据)。对于表达分析,使用了Cosmic财团的数据(使用Affymetrix HT-HG-u133av2平台对789个癌细胞系进行基因表达分析,来源:EBI ID:E-MTAB-78322)。
在n维向量的集合中,这些非零向量(具有至少一个非零分量)之间的相似性可以通过计算内积空间的余弦α来确定,或者换句话说,特定药物的药物响应向量与所有其他药物响应向量的夹角。对于所有计算,所有细胞系的药物剂量-反应数据之间的关系计算为共亨距离49,50。聚类树中两个对象的共亨距离是分隔两个对象的分支的深度,由以下公式(式(1))定义:  GDSC百科全书的AUC值(仅MGH数据,另见下文“质量控制:数据集质量”)以每细胞系1-AUC的形式存储在分类数据向量中。AUC值在0和1之间波动;0反映最高响应。药物A与药物B之间的距离计算为1减去AUC数据向量之间的相似性。剂量-反应数据采用Ward.D2或平均聚类法进行聚类。根据我的癌症基因组,使用本体组计算路径内距离,来源见补充数据1。排除已知跨多条通路交叉反应的药物,计算通路内距离。
GDSC百科全书的AUC值(仅MGH数据,另见下文“质量控制:数据集质量”)以每细胞系1-AUC的形式存储在分类数据向量中。AUC值在0和1之间波动;0反映最高响应。药物A与药物B之间的距离计算为1减去AUC数据向量之间的相似性。剂量-反应数据采用Ward.D2或平均聚类法进行聚类。根据我的癌症基因组,使用本体组计算路径内距离,来源见补充数据1。排除已知跨多条通路交叉反应的药物,计算通路内距离。
为了生成药物图谱Vonoroi图,将GDSC-MGH剂量反应实验的AUC转换为余弦α向量。所有药物对所有药物的距离矩阵采用层次聚类算法进行聚类,该算法采用欧几里德距离度量和两两平均连锁,产生一个基于药物在不同细胞系中的反应分离药物的聚类树。该二叉聚类树随后使用加权快速Voronoi布局(WFVL)算法的自适应版本绘制为平坦地图。在Voronoi图中,根据平面上的一组位置将平面划分为多个区域。在两个站点之间的距离相等的地方绘制区域之间的边界。这将产生一组多边形。在加权Voronoi图中,根据站点的权重计算到站点的距离。质心Voronoi图通过考虑多边形边的宽高比来移动位置,以获得平面上位置的均匀分布。通过递归地将加权质心Voronoi图应用于数据的聚类树,我们可以将该数据结构映射到一个平面上(另请参见补充图1a)。
因为这是一个不确定的算法,所以需要设置阈值来完成计算。此外,这些计算是计算密集型的,因此需要启发式算法在可接受的时间内计算。WFVL中最重要的启发式是使用“功率图”;将二维平面转换为三维凸包,从而可以快速计算质心。
使用的其他启发式方法是在内部开发的,并在下面的伪代码中描述: 1 基于在树深度d处形成的百分比簇计算分支分组阈值→分支阈值b应用启发式算法 2 分支距离低于阈值b时分组 3 基于相似性识别基因的早期分裂→分组聚类树 4 递归布局分支:对每个分支应用加权快速Voronoi布局(FVL)→近似Voronoi映射Va 5 通过重新应用FVL平滑Va映射,同时保持相对位置→均匀分布的Voronoi映射V 区域之间的边界厚度反映了聚类树中的截止值(同系词距离);越高,边界越厚。
基于灵敏度、距离和突变的协同预测模型。该模型不仅利用不同细胞系对单个药物的敏感性数据,而且利用药物图谱距离预测药物协同效应。使用和不使用药物图谱距离进行预测,以证明其附加值。对于数据准备,数据中的每一行对应一个细胞系和一对药物的值。因此,通过药物对,数据中有很多结构。除了药物图谱中两种药物之间的距离外,数据还包括个体药物敏感性值以及与两种药物相关的靶向信息,其中靶向信息表明药物是否靶向已知受影响/突变的细胞系基因。最后,如果已知这两种药物显示协同作用,它还包括一个指标变量。对于每个药物对和细胞系,首先提出目标函数(式(2))
 模型的优化。为了预测两种药物是否具有协同效应,我们分析了以协同效应指标为响应的logistic回归模型,该模型的协变量包括(1)个体药物敏感性,(2)靶点信息和(3)药物距离。请注意,药物协同作用信息保留为0的行可能未知或没有协同作用。我们知道协同变量θij取决于药物图谱上药物对(i,j)之间的距离dij,以及靶信息tijk,这表明是否至少有一种药物靶向细胞系k中突变的基因。我们可以将i,j索引的两种药物之间的协同作用写成(等式(3)):
模型的优化。为了预测两种药物是否具有协同效应,我们分析了以协同效应指标为响应的logistic回归模型,该模型的协变量包括(1)个体药物敏感性,(2)靶点信息和(3)药物距离。请注意,药物协同作用信息保留为0的行可能未知或没有协同作用。我们知道协同变量θij取决于药物图谱上药物对(i,j)之间的距离dij,以及靶信息tijk,这表明是否至少有一种药物靶向细胞系k中突变的基因。我们可以将i,j索引的两种药物之间的协同作用写成(等式(3)):  逻辑回归。数据包含一个指标变量标签,对于两种药物显示协同作用的情况,该标签为1,否则为0。对于每个组织,我们使用标签为1的所有行拟合逻辑回归,然后为标签为0的组织随机选择相同数量的细胞系(观察)。
逻辑回归。数据包含一个指标变量标签,对于两种药物显示协同作用的情况,该标签为1,否则为0。对于每个组织,我们使用标签为1的所有行拟合逻辑回归,然后为标签为0的组织随机选择相同数量的细胞系(观察)。  药物敏感性与协同效应的关系。为了将协同效应的发生与药物敏感性相关联,我们对每种组织类型每种药物的药物敏感性进行标准化,以校正组织特异性动态范围。为此,将每种药物每种组织类型的敏感性转移到标准化平均值,该值被设置为1(每种药物每种组织的平均2log IC50浓度)。与标准化值相比,显示的所有灵敏度均为δ2log IC50。
药物敏感性与协同效应的关系。为了将协同效应的发生与药物敏感性相关联,我们对每种组织类型每种药物的药物敏感性进行标准化,以校正组织特异性动态范围。为此,将每种药物每种组织类型的敏感性转移到标准化平均值,该值被设置为1(每种药物每种组织的平均2log IC50浓度)。与标准化值相比,显示的所有灵敏度均为δ2log IC50。